E. coli (EC) – culture et sérotyypage
E.coli est une bactérie gram-négative en forme de bacille qui a une multitude de variants. En plus de souches commensales qui se trouvent naturellement dans la flore intestinale de tous les mammifères, il existe aussi des souches pathogènes classées comme E.coli intestinal (InPEC) ou extra-intestinal (ExPEC). Cependant, la différenciation de ces variants a soulevé un problème de diagnostic pendant longtemps à cause des différents pathotypes ou pathovars qui ne peuvent pas être identifiés avec des méthodes phénotypiques simples.

E. coli peut être détecté chez les porcs avec une relative facilité par culture sur gélose au sang (figure 1) ou des milieux sélectifs (par exemple gélose Gassner ou MacConkey) d’échantillons adéquats (intestin grêle, fèces) d’animaux avec une infection aigüe. Auparavant, on avait utilisé avec succès le typage des sérotypes ou sérovars pour différencier les isolats d’E.coli car certains sérotypes sont fréquemment associés à des maladies déterminées (tableau 1). Pour cela, en vétérinaire, il est courant de déterminer les antigènes O et F par agglutination rapide en utilisant des sérums poly ou monovalents (les antigènes O sont des composants des LPS de la paroi cellulaire bactérienne et sont aussi endotoxiques ; les antigènes F (fimbriaux) sont des adhésines qui facilitent l’adhésion à l’hôte- spécifique pour coloniser les cellules épithéliales de l’intestin grêle). Bien que cette méthode soit rapide, elle est sujette à des erreurs par des réactions croisées, masquage des antigènes cibles ou incapacité des fimbriae à se développer in vitro, ce qui peut conduire à des classifications erronées. Par ailleurs, depuis des décennies la séroformule O:K:H a été et continue d’être la norme pour une détermination complète de tous les sérotypes, par exemple pour déterminer non seulement les antigènes O mais aussi les antigènes K (polysaccharide capsulaire) et H (protéine flagellaire).
Tableau 1 : Pathogènes E.coli intestinaux chez les porcs
| Pahotypes |
Maladies dues à E. coli |
Sérotypes d’antigène O fréquemment associés | Sérotypes fimbriaux fréquemment associés |
| E. coli pathogène intestinal (InPEC) | Diarrhée à E. coli chez les porcelets | O8, O108, O138, O139, O141, O147, O149 et O157 | F4, F5, F6 y F41 |
| Diarrhée à E. coli chez les porcs sevrés | O8, O108, O138, O139, O141, O147, O149 et O157 | F4, F18ac | |
| Maladie de l’œdème chez les porcs sevrés | O138, O139 et O141 (O147, O157 et régulièrement des non sérotypables) | F18ab |
E. coli (EC) – pathotypes et virotypage
D’autres avancées dans les méthodes de biologie moléculaire et des améliorations dans l’analyse biologique moléculaire d’E. coli ont conduit à l’identification de beaucoup de gènes qui codent des facteurs de virulence (tableau 2) : d’une part, des facteurs de virulence classiques avec des effets nocifs (toxines) et, d’autre part, ceux qui directement ou indirectement facilitent la colonisation et la survie d’E. coli chez l’hôte. L’identification de ces gènes, par exemple, par PCR ou technique array, fait que maintenant il est possible de déterminer la virulence potentielle des isolats de E. coli et de faire la distinction entre différents pathotypes (tableau 3) L’identification de ces marqueurs de virulence et par conséquent de leurs gènes cible s’appelle le virotypage.
Tableau 2 Exemples de gènes cibles détectés sur E. coli et les facteurs de virulence (fimbriae, adhésines, toxines) pour lesquels ils codent.
| Gènes cibles | Facteurs de virulance |
| faeG (F4) | Fimbriae F4 |
| fanC (F5) | Fimbriae F5 |
| fasA (F6) | Fimbriae F6 |
| fedA (F18) | Fimbriae F18 |
| fim41A (F41) | Fimbriae F18 |
| fimH (F1) | Fimbriae type 1 |
| fimA (F1) | Fimbriae type 1 |
| papC | Fimbriae P |
| aidA (AIDA) | Adhésine auto transporteuse AIDA-I |
| paa | Facteur d’adhérence porcin |
| eaeA (intimine) | Intimine |
| eltB (LTI) | Entérotoxine sensible à la chaleur |
| estA (STI) | Entérotoxine résistante à la chaleur |
| est B (STII) | Entérotoxine résistante à la chaleur |
| estA (EAST) | Entérotoxine résistante à la chaleur |
| stx2e | Shigatoxine variante 2e |
| cdtB | Cytolethal distending toxin |
| cnf1 | Facteur cytotoxique nécrosant de type 1 |
| iucD | Aérobactine |
| escV | Système de sécrétion de type III |
| pic | Serin protease autotransporter |
Tableau 3 : Gènes cibles pour l’identification de pathotypes d’E. coli chez les porcs
| Pathotypes | Fimbriae et adhésines (gènes cibles) | Toxines (gènes cible) | Symptômes | |
| E. coli pathogène intestinal (InPEC) |
E. coli entéropathogéne(EPEC)
|
fimA/fimH paa intimine |
escV pic cdtB |
Diarrhée par malabsorption |
|
E. coli entérotoxique (ETEC)  |
F4, F5, F6, F18, F41 fimA/fimH AIDA paa |
STI, STII, EAST LTI Stx2e hémolysine |
Diarrhée par hypersécrétion | |
| Maladie de l’œdème par E. coli (EDEC) | F18 AIDA |
Stx2e hémolysine |
Maladie de l’œdème | |
| E. coli formatrice de Shigatoxine (STEC) | fimA, fimH intimine |
Stx2e EAST escV pic, cdtB hémolysine |
Diarrhée catarrhale hémorragique | |
| E. coli patógeno extraintestinal(ExPEC) |
E. coli septicémique (SEPEC) |
papC | iucD | Septicémie hémorragique |
| E. coli uropathogéne(UPEC) | papC fimA, fimH | cnf1, iucD hémolysine |
Mammite par coliformes sur les truies | |
Cependant, les isolats d’E. coli pathogène contiennent toujours une combinaison de fimbriae et d’adhésines en plus des toxines. Plusieurs toxines et adhésines peuvent être présentes à la fois et dans différentes combinaisons. La détermination de ces différents marqueurs de virulence et l’identification de leurs gènes fournit l’information sur le pathotype d’E. coli isolé de la culture cellulaire (tableau 3) Il faut souligner que les E. coli hémolytiques ne sont pas les seuls à être pathogènes (figure 1) et de fait, les E. coli non hémolytiques en particulier, sont fréquemment déterminés comme responsables de maladie clinique. D’autre part, Par conséquent, si c’est possible, tous les variants d’isolats d’E.coli identifiés par culture doivent être virotypés afin de s’assurer que le pathogène responsable peut aussi être identifié. L’information sur les types et les toxines fimbriaux d’E. coli pathogènes déterminés est aussi utile dans le choix de vaccins adéquats avec les antigènes correspondants.
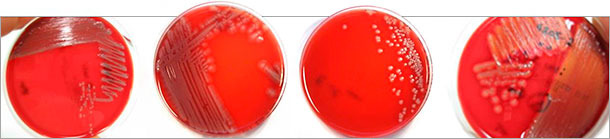
Figure 1 : Différentes morphologies coloniales d’isolats d’E. coli pathogènes sur gélose au sang (Colombie) identifiés après virotypage par PCR multiple. Les E. coli hémolytiques (à droite) sont pathogènes, mais pas seulement.
Dans l’interprétation des résultats de diagnostic, il faut tenir compte que les pathotypes d’E. coli peuvent aussi être isolés chez des hôtes sains (infection latente) surtout de la flore intestinale. Par conséquent, l’évaluation des résultats ne peut seulement être faite qu’en prenant en compte les découvertes cliniques chez l’animal ainsi que la concentration des pathogènes isolés avec le pathotype identifié.
L’interprétation du virotypage devient compliquée quand les marqueurs de virulence sont directement détectés dans le matériel de l’échantillon, par exemple les fèces, sans besoin de typer les isolats individuels. Bien que ceci soit techniquement possible et on le réalise sur demande, quand différents pathotypes d’E. coli sont présents dans l’échantillon, on obtient aussi un mélange de tous les facteurs détectables qui peut conduite à l’identification de faux pathotypes et une combinaison de marqueurs de virulence qui n’existe pas comme telle. De plus, il est aussi possible qu’on détecte des marqueurs de virulence d’autres bactéries intestinales présentes dans l’échantillon de fèces car ces bactéries ont des gènes semblables. Dans ce cas, on détectera d’éventuels marqueurs de virulence d’E. coli qui, de fait, ne sont pas des facteurs de virulence pour l’E. coli détecté dans l’échantillon. Par conséquent, on doit porter une attention soignée dans l’interprétation des résultats obtenus dans le virotypage des échantillons.
En combinaison avec l’examen clinique et l’examen pathologique souvent nécessaire (décrit auparavant) et avec les diagnostics différentiels appropriés, le virotypage des isolats d’E. coli fournit une nette amélioration dans le diagnostic des infections par E. coli chez le porc.



