La révolution génomique a été possible grâce aux technologies de séquençage connues comme de "nouvelle génération" ou à "haute performance", qui ont été ébauchées dans le récent article "Que veut-on dire par révolution génomique ? »
Les principes de la sélection génomique sont :

- L'ADN est analysé au moyen d'une puce pour identifier des différences dans le code génétique entre animaux appelées polymorphismes de nucléotide unique ("Single Nucleotide Polymorphisms", SNP).
- Les SNP's mettent en rapport des segments spécifiques de l'ADN avec des différences productives pour des

L'un des potentiels à long terme de la sélection génomique est l'identification de gènes spécifiques ou régions génomiques avec un impact significatif sur la santé porcine et permettant d'améliorer la sélection pour la résistance et/ou la tolérance aux maladies. Quelques grands projets et collaborations de recherche travaillent sur ces secteurs et leurs résultats vont élargir la base de données existante dans la littérature scientifique.
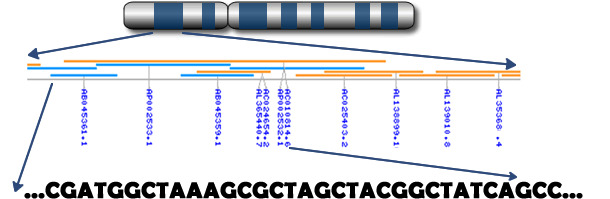
Figure 2. La génomique identifie les SNPs pour les caractères de santé (Source: Prof. C. Haley – Roslin Institute, Edinburgh)
Par exemple, dans l'édition récente de l'influente PAG (International Plant and Animal Genomics) au début de cette année, parmi les principaux articles présentés on trouvait les suivants :
Des chercheurs des USA ont identifié trois marqueurs qui sont significativement associés avec des lésions pulmonaires dues à la pleuropneumonie et/ou la pneumonie enzootique. On espère que ceci permettra d'identifier la variation génétique en rapport avec l'augmentation de la résistance aux maladies respiratoires.
- Des scientifiques chinois ont travaillé sur les mécanismes qui expliquent la différence sur l'état sanitaire entre les porcs Landrace (sensibles) et Tongcheng (une race chinoise, qui est résistante au SDRP). Ils ont observé l'expression différenciée du gène CD169 chez les porcs Tongcheng par rapport aux Landrace après l'infection par le SDRP. Quelques études présentées montrent que le récepteur CD169 améliore la présentation de l'antigène aux cellules T.
- Le SDRP Host Genetics Consortium (PHGC) a été créé par des groupes de recherche du Canada et des USA pour déterminer le rôle de la génétique dans la résistance à l'infection face au SDRP et les effets en rapport avec la croissance. Les derniers résultats d'études sur des porcelets en post-sevrage ont identifié une région génomique dans le chromosome 4 (SSC4) avec un impact significatif sur la variabilité de la charge virale et la croissance après l'exposition au virus. Des analyses postérieures et le séquençage de la région SSC4 devraient identifier des marqueurs qui permettent de différencier les animaux résistants au SDRP /avec une croissance maximale et les animaux sensibles au SDRP/avec une croissance réduite.
- D'autres projets du PHGC étudient le transcriptome des porcs exposés au SDRP. Pour que les gènes produisent des protéines, l'ADN est transcrit à un ARN messager (mRNA). Le terme transcriptome comprend tout le mRNA qui est utilisé pour la production de protéines. Cette étude fournira davantage de données pour déchiffrer les mécanismes génétiques de la réponse de l'hôte face à l'infection par le SDRP.
- Des chercheurs de l'Iowa State ont présenté une analyse génomique qui a révélé des différences d'expression dans beaucoup de gènes entre des porcs classés comme excréteurs de salmonella persistants ou modérés. Ils ont conclu que les différences quantitatives dans les valeurs d'IFN-γ expliquent l'expression de la majorité des gènes analysés, et que le régulon IFN-γ est une source de gènes dont le niveau d'expression deux jours après l'infection peut prédire la capacité excrétrice de l'animal. Ces gènes peuvent être évalués comme candidats pour le développement d'analyses prédictives sur la capacité excrétrice des animaux.
- Des chercheurs chinois ont étudié la réponse immunitaire sur des porcelets pour 18 caractères hématologiques, 7 lymphocytaires et 3 des cytokines en utilisant un vaccin contre la peste porcine sur deux génotypes différents. Ils ont identifié 12 SNPs significatifs reliés avec la réponse immunitaire, ce qui suggère une base préliminaire pour une identification postérieure des mutations causales sous-jacentes à la réponse immunitaire générale.
- Des chercheurs du Nebraska ont travaillé avec des races croisées infectées expérimentalement par le PCV2b pour identifier les principales variations génétiques qui influencent les principaux indicateurs de la progression de la maladie et de la réponse immunitaire contre le PCVAD. Pendant une période de 28 jours d'exposition au virus, on a mesuré chaque semaine le GMQ, la charge virale et les valeurs d'anticorps spécifiques. Les premiers résultats ont montré une variation entre les hôtes quant à l'ampleur et au temps de réponse immunitaire contre le PCV2b. Les principaux clusters de SNPs qui ont affecté la charge virale étaient localisés dans de multiples chromosomes comprenant SSC6, SSC7 et SSC12. La région située en SSC7 a aussi touché le gain de poids pendant l'exposition. Le cluster de gènes pour l'antigène leucocytaire porcin classe II est localisé dans cette région et peut expliquer en grande partie la variation phénotypique observée.

Figure 3. PCVAD – Un futur objectif clé pour la sélection génomique (Photo: J. Mackinnon).
Outre les études citées, on fait des recherches sur les gènes/SNPs impliqués dans d'autres maladies significatives comme la fièvre aphteuse, la PPA et la grippe porcine. Toutefois, la sélection future pour une amélioration de la santé demandera l'utilisation de multiples marqueurs et la disponibilité d'importantes bases de données d'ADN de plusieurs populations bien caractérisées tant génétiquement que phénotypiquement dans lesquelles on peut identifier des associations entre les SNPs et les caractères de santé. Le prochain article de cette série, « Quand la génomique portera-t-elle ses fruits ?" analysera ceci avec plus de détails.