Le PCV2 a émergé comme l'une des infections virales les plus dévastatrices en production porcine, ce ayant un impact économique important pour les pertes directes et les dépenses en stratégies de contrôle. Le PCV2 est l'agent étiologique du syndrome d’amaigrissement du porcelet en PS (MAP), aujourd'hui connu comme maladie systémique due au PCV2 (MS-PCV2, Figure 1). Les études épidémiologiques et expérimentales ont montré que la diversité génétique peut affecter potentiellement la virulence de PVC2, caractérisée par des vagues mondiales de génotypes émergents et la circulation de souches recombinantes. Peut-être cela est la raison pour laquelle la classification intraspécifique du PCV2 a toujours été controversée. En 2008, le consortium européen des maladies associées au circovirus porcin a proposé une nomenclature normalisée pour les définitions génotypiques du PCV2 basées sur la comparaison des séquences appariées (www.pcvd.eu). L'analyse des séquences de nucléotides du génome de PCV2 complet et de la capside (ORF2) ont défini deux seuils de distance de 0,020 et 0,035, respectivement. Lorsque la distance (p) entre deux séquences est supérieure à ces valeurs, on a supposé que les souches appartenaient à deux génotypes différents. Les résultats de ces analyses ont reconnu quatre génotypes appelés PCV2a, PCV2b, PCV2c et PCV2d (également connu sous le nom PCV2b mutant).


Figure 1. Porc de 3 mois atteint de MS-PCV2. Notez la colonne vertébrale marquée, indicatrice d'un retard de croissance , et la pâleur (anémie).
Depuis 2008, un grand nombre de séquences de PCV2 ont été déposées dans la GenBank (www.ncbi.nlm.nih.gov) et on a proposé plusieurs nouveaux génotypes, même si ils n’ont pas toujours été validés. Une proportion significative de ces séquences a une origine recombinante et dans certains cas a recirculé avec une augmentation de la prévalence dans plusieurs pays d'Asie et aux États-Unis. Dans un article récent (Franzo et al, 2015), une équipe internationale de chercheurs a réanalysé la taxonomie intraspécifique du PCV2 et la définition génotypique pour vérifier leur validité actuelle, unifier la nomenclature et éviter d'autres interprétations erronées.
Les résultats obtenus indiquent que, avec les séquences actuellement disponibles de capside et de génome de PCV2, plusieurs des hypothèses de la méthodologie qui a été créé il y a près d'une décennie pour établir les seuils ont été violées. Plus précisément, l’hypothèse du même taux de substitution de nucléotides entre les différents génotypes de PCV2 n'a pas été respectée. En outre, la définition d'une seule valeur de coupe pour définir les génotypes de PCV2 semble compliquée. Plus important encore, les seuils proposés pour définir le génotype manqueraient sans aucun doute de sens (figure 2). Ces évidences, ainsi que l'énorme quantité de nouvelles séquences disponibles, ont des implications importantes pour la classification intraspécifique du PCV2. Par conséquent, les seuils appliqués à partir de 2008 pour définir les génotypes de PCV2 ne sont pas applicables à toutes les souches de PCV2 et la méthode doit donc être reconsidérée.
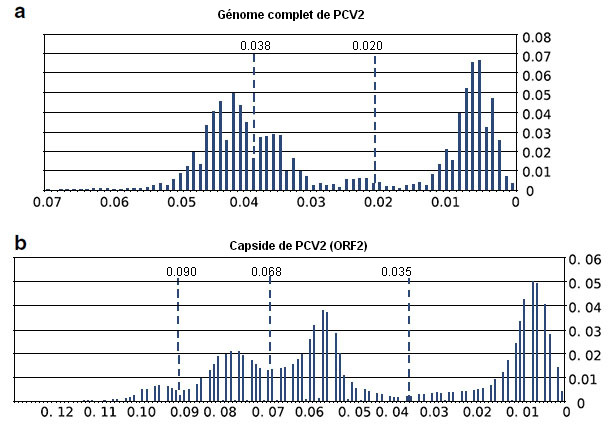
Figure 2. Résultats de la comparaison de séquences du génome complet du PCV2 (a) et de séquences d'ORF2 (b). On représente la proportion de séquences comprises dans un intervalle de distance p de 0,01.Sont aussi indiqués les seuils proposés pour le génome complet (0,020), la capside (0,035), ainsi que les seuils résultant des réanalyses réalisées (0,038, 0,068 y 0,090).
En conséquence, une méthode alternative est proposée afin de déterminer sans équivoque le génotype des souches de PCV2. La méthode proposée tient compte de la présence de plusieurs souches recombinantes qui, en fait, appartiennent à plus d'un génotype. Compte tenu du taux élevé de recombinaison décrit, on a préféré une classification basée sur le gène ORF2. Dans le but de fournir une méthode de classification non ambiguë, plusieurs séquences de référence dont la classification était claire selon les méthodes phylogénétiques ont été sélectionnées. Cela nous a permis de définir quatre génotypes utilisant à la fois la reconstruction phylogénétique et quarante-sept positions de nucléotides marqueurs spécifiques pour chaque génotype. Ces positions de marqueurs caractérisaient chaque génotype d'une manière cohérente (> 95%) et pouvaient être utilisés comme référence pour l'attribution d'une séquence à problème à un génotype déterminé. La méthode de classification proposée, en plus d'être robuste, est très rapide et facile à réaliser.
La classification taxonomique des PCV2 reste un défi. Les résultats obtenus confirment et valident la variabilité des séquences virales et la fréquence élevée de recombinaison intra- et inter-génotype entre les souches de PCV2, mettant en évidence la difficulté d'établir une définition claire du génotype pour ce virus. En outre, la possibilité de l'apparition dans le futur de nouveaux génotypes du PCV2 est très élevée et il peut être beaucoup plus nécessaire de disposer de nouvelles et robustes méthodes de génotypage.
La classification des génotypes de PCV2 est importante d'un point de vue pratique. En outre, tous les vaccins disponibles sur les marchés européens et nord-américains sont basés sur le génotype PCV2a, bien que ce soient les PCV2b et PCV2d les plus répandus. Bien qu'on ait montré un niveau significatif de protection croisée entre ces trois génotypes il serait intéressant d'évaluer si l'efficacité du vaccin est équivalente contre tous ces génotypes. En outre, jusqu'à présent, une étude phylogénétique publiée indique que certains changements dans les séquences d'acides aminés de PCV2 issus d’élevages vaccinés et non vaccinés peuvent se produire. En fait, il a été spéculé sur la possibilité que des virus mutants soient générés en raison de la pression de la vaccination. Il suivi est donc impératif de mettre en place un suivi génotypique approprié pour évaluer l'efficacité du vaccin.




