Avec la collaboration de Joan Lunney (ARS USDA) et Jack Dekkers (Iowa State University), le SDRP Host Genetics Consortium (PHGC) a été mis en place pour introduire des outils génétiques et génomiques pour minimiser l’impact du SDRP. L’objectif est de combiner les technologies de dernière génération en virologie-immunologie et en génétique-génomique. La principale prestation est l’application des résultats dans des programmes commerciaux de reproduction. Les premiers essais pour identifier les gènes qui induisent de la résistance aux maladies ont été centrés sur la comparaison de la réponse face aux maladies des porcs de différentes lignées pures. Le PHGC ne fait pas de comparaison entre les races mais il se concentre sur l’identification de marqueurs de résistance/sensibilité présents dans les populations d’animaux commerciaux qui présentent divers phénotypes de maladie.
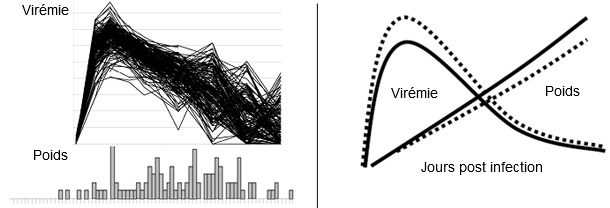
En suivant ce modèle, on a infecté expérimentalement 200 porcelets avec des souches bien caractérisées de virus SDRP. On a récolté des données individuelles sur le phénotype de la maladie : mortalité, charge virale dans le sang, GMQ, valeurs d’anticorps neutralisants et de cytokine sérique (voir figure 1). Le génotypage de tous les porcs avec le chip porcin 60KSNP et la réalisation de larges études d’association génomique ont servi pour identifier différents marqueurs en relation avec la progression de la maladie clinique et sa résistance. L’allèle favorable sur un marqueur important dans le chromosome 4 du porc (Sus scrofa chromosome 4, SSC4) est associé à une augmentation du GMQ et une diminution de la charge virale. Pendant les 42 jours de l’infection expérimentale, les porcs avec le génotype favorable ont gagné 10% de poids en plus et ont montré une diminution de la virémie. L’effet du génotype bénéfique est modélisé sur la figure. Les résultats jusqu’à ce jour montrent qu’un bénéfice de l’allèle favorable similaire peut être obtenu en conditions de terrain.

|
Figure 1 : Phénotypes de maladie causée par le virus SDRP au niveau de la population. Les figures montrent la virémie du virus SDRP (figure supérieure) et la répartition du GMQ (inférieure) sur 200 porcs infectés expérimentalement avec le virus SDRP. Les porcs ont été infectés expérimentalement à 3 semaines de vie et on a récolté les données 42 jours après l’infection. |
Figure 2 : L’effet favorable du marqueur bénéfique dans le SSC4 sur le GMQ et sur la virémie chez de jeunes porcs. Le modèle supérieur montre les améliorations attendues sur le GMQ et sur la virémie avec l’allèle favorable (AB ou BB, ligne solide) face à des porcs avec le génotype AA (ligne de points). L’augmentation du GMQ est d’environ 10%. La diminution du pic de virémie est d’environ 1 log. |
On a détecté des résultats phénotypiques supplémentaires qui étaient en relation avec différents aspect de l’immunité spécifique contre le SDRP. Par exemple, on a identifié une relation inverse entre la charge virale et les titres neutralisants contre le virus. De plus, un faible pourcentage de porcs infectés a produit une réponse d’anticorps unique, qui peuvent être identifiés comme largement neutralisants et qui avait la capacité de neutraliser une large gamme de souches y compris les virus de type 1. Bien que l’on n’ait pas détecté de composante héréditaire pour la réponse immunitaire, le niveau d’anticorps anti-protéines N contenait, lui, un composant héritable spécifique, en relation avec un SNP dans le SSC7. Un marqueur dans le SSC1 est en corrélation avec différents problèmes respiratoires chez les porcs avec du CRP. Le marqueur génétique plus proche est proche d’un récepteur de cytokines inflammatoires.
Les résultats du travail mené par le PHGC illustrent d’importantes découvertes. Premièrement, la maladie clinique et les conséquences de l’infection par le SDRP ont une composante héréditaire significative. Deuxièmement il y a un nombre relativement faible de marqueurs génomiques associés à un phénotype déterminé de maladie. Il est donc possible d’envisager une sélection assistée par des marqueurs pour produire des porcs avec une meilleure réponse à l’infection. Et enfin, dans la mesure où tous les marqueurs génomiques en relation avec la maladie clinique et l’immunité se trouvent dans des régions du génome porcin avec des gènes immunologiquement importants, y compris des gènes stimulés par interféron, des gènes CMH et des gènes qui répondent à la cytokine.
En conclusion, la génomique et la génétique sont des outils pour améliorer la santé animale, particulièrement dans les maladies infectieuses qui n’ont pas de vaccins ou de traitements efficaces. Quand on combinera l’amélioration génétique avec de meilleurs vaccins et une meilleure alimentation, il sera possible d’inverser la plus grande partie des effets négatifs du SDRP endémique.