Les virus de l'influenza porcin (SIV ; sigles en anglais) font partie du genre Influenzavirus type A de la famille Orthomyxoviridae. Ces virus sont sous-classifiés en fonction des glycoprotéines de surface : l'hémagglutinine (HA) et la neuraminidase (NA), dont 17 et 10 types ont été décrits respectivement. La combinaison d'un type d'HA et d'un type de NA est à la base de ce que l'on appelle sous-type. Ainsi, chez le porc, les sous-types majoritaires sont H1N1, H1N2 et H3N2.
Les différentes études réalisées en Europe permettent d'affirmer que la séroprévalence des trois sous-types (H1N1, H1N2 et H3N2) est très élevée dans les pays où la production porcine est importante. Par exemple, en Allemagne, en Belgique, au Danemark ou en Espagne, la séroprévalence des trois sous-types dépasse 30%. En Espagne, plus particulièrement, 94% des exploitations sont positives à au moins un sous-type. En France ou au Royaume-Uni, on a détecté des prévalences élevées d'H1N1 ou d'H1N2 ; cependant, il semble que le sous-type H3N2 n'y est pas présent, au moins ces dernières années. Dans les pays où la production est plus faible, comme en République Tchèque, en Irlande ou en Pologne, la prévalence des SIV semble être inférieure.

Dans une même exploitation, il est possible de détecter de manière directe (par PCR) ou indirecte (par inhibition de l'hémagglutination) plusieurs sous-types de SIV circulants. En effet, c'est une situation qui a été constatée dans des exploitations aux Pays-Bas, en France ou en Espagne, parmi d'autres. À titre individuel, il faut dire aussi que la proportion d'animaux présentant des anticorps face à plusieurs sous-types peut être élevée autant chez les truies reproductrices que chez les porcs en engraissement. Ce fait suggère que l'infection par SIV est active et fréquente. Malgré cela, la présence du virus n'est pas toujours associée à des poussées de pathologie respiratoire ; en effet, il existe un nombre élevé d'exploitations positives au virus sans signes cliniques apparents. En définitive, ces découvertes suggèrent que le SIV se présente de manière importante comme une infection enzootique.
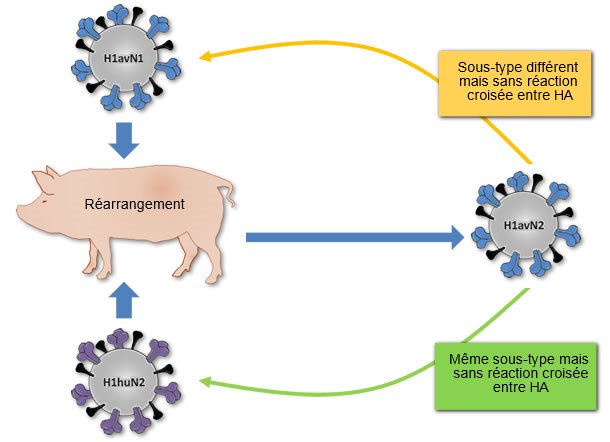
Figure 1. Origine du virus H1avN2 décrit au Danemark et en France. Ce virus est généré à partir du réarrangement des virus H1avN1 (apportés par l'HA) et le virus H1huN2 (apporté par la NA). Comme le virus H1huN2, l'H1avN2 fait partie du sous-type H1N2 ; cependant, son HA ne réagit pas de manière croisée avec ce virus, mais si avec l'HA du virus H1avN1, même si celui-ci appartient à un sous-type différent (H1N1).
La classification par sous-types est utile pour simplifier et comprendre l'épidémiologie et la diversité des virus de l'influenza ; cependant, la diversité des SIV est, à vrai dire, plus complexe. Comme le Dr. Simon le résume très bien dans un autre article publié sur ce même site web, au moins 4 lignées principales peuvent être distinguées en Europe en fonction du type d'HA qu'elles contiennent. Ainsi, on peut distinguer celles en rapport avec les H1N1 de type aviaire (H1avN1), celles en rapport avec les H1N2 (H1huN2), celles en rapport avec les H1N1 pandémiques (H1N1pdm) et finalement celles en rapport avec les H3N2.
Force est de rappeler que les SIV, comme tout autre virus appartenant aux influenzavirus A, sont composés par un génome segmenté en 8 morceaux d'ARN, ce qui leur permet d'évoluer moyennant deux mécanismes : 1) la dérive antigénique ; des mutations qui, produites à des endroits spécifiques de l'HA (essentiellement) ou de la NA, peuvent générer des variantes virales qui échappent à l'immunité générée préalablement dans la population par des virus ancêtres (cause principale de la grippe saisonnière chez l'être humain), et 2) le réarrangement génétique, qui se produit lorsque deux ou plusieurs virus échangent des morceaux d'ARN (par exemple, l'apparition en 1994 de la souche H1huN2, résultante du réarrangement entre un H1N1 humain et l'H3N2 porcin).
Bien que les trois H1 différents (H1avN1, H1huN2 et H1N1pdm) aient partagé dans le passé un ancêtre commun, il n'y a pas de croisement entre eux au niveau antigénique, ou alors il est très limité. Ceci est probablement dû au fait que les trois H1 ont évolué par dérive antigénique de manière isolée dans trois espèces différentes avant leur introduction dans le porc en Europe : dans des oiseaux l'H1avN1, dans des humains l'H1huN2, et dans des porcs en Amérique du Nord l'H1N1pdm. Par ailleurs, le réarrangement génétique peut générer de nouvelles souches de virus de l'influenza qui, même si elles appartiennent à un même sous-type, peuvent faire l'objet d'un croisement antigénique avec des virus appartenant à d'autres sous-types. La souche H1avN2 décrite au Danemark et en France en est un bon exemple. Cette souche contient une HA en rapport avec celle présente dans les souches H1avN1 et, par conséquent, il n'y a pas de croisement antigénique avec la souche H1huN2, mais si avec les souches H1avN1.
La diversité génétique et antigénique des virus de l'influenza doit être comprise comme étant dynamique et en évolution permanente. Les travaux récemment publiés, réalisés en Allemagne, au Danemark, en Espagne ou en Italie, ainsi le confirment. Dans ces travaux, neuf souches issues du réarrangement entre les souches de SIV porcines et des souches de la grippe saisonnière humaine ont été isolées. Pour toutes ces raisons, il est extrêmement important de maintenir et même de renforcer une surveillance épidémiologique active des SIV afin de connaître les souches présentes dans le cheptel porcin, ainsi que leur prévalence et leur impact.




