Introduction
Le génome du virus du SDRP est formé par une simple chaîne d’ARN ce qui le rend très sujet aux mutations génétiques. Cela fait aussi que chaque souche de SDRP est unique, c’est pourquoi le typage génétique est une méthode utile pour le diagnostic et le contrôle de la maladie. Le diagnostic par typage génétique se fait en déterminant l’ordre des nucléotides à l’intérieur d’une copie d’ADN d’un fragment du génome ARN par le séquençage d’ADN. Actuellement, le fragment le plus utilisé est l’ORF5, le gène qui code pour la glycoprotéine plus importante de l’enveloppe, principalement parce qu’il montre une grande diversité génétique.

Diagnostic par séquençage d’ADN
La discrimination entre le SDRP de type 1 (européen) et de type 2 (américain) se fait facilement avec la plupart des diagnostics de PCR mais la discrimination entre des souches individuelles à l’intérieur de chacun des deux génotypes demande un séquençage d’ADN. Pour cela, on utilise les fluides corporels ou les tissus avec une charge entre modérée et sévère de SDRPv, desquels on isole l’ARN qui est copié vers l’ADN par une transcription inverse. On amplifie alors le gène ORF5 par PCR et on envoie dans un laboratoire de séquençage d’ADN. Ce procédé est très automatisé et demande entre 1 à 3 jours. Les données brutes du séquençage sont envoyées au laboratoire de diagnostic pour leur analyse. Le rapport comprend normalement le séquençage des nucléotides de la souche, leur similitude par rapport aux souches vaccinales standard et quelques laboratoires fournissent une comparaison avec un tableau de référence d’isolats sauvages de SDRPv en forme de dendrogramme ou la comparaison avec la base de données de SDRPv d’un système de production.
Analyses de séquences de SDRPv
La similitude ou l’identité entre séquences sont déterminées en alignant deux séquences ou plus en utilisant un programme informatique. La figure 1 représente un exemple d’alignement de séquences d’ORF5 de 5 élevages différents. Les comparaisons par paires du pourcentage d’identité sont montrées sur le tableau 1. Les identités oscillent entre 81,2 et 99,8%. Le dendrogramme découlant de l’analyse phylogénétique montre un groupement des séquences similaires (figure 2). Une question clé pour les producteurs et les vétérinaires est de savoir si les différentes génétiques observées entre les séquences représentent une variation normale d’une même souche de SDRPv de l’élevage ou représentent plusieurs souches distinctes sur le même élevage.

Figure 1 : fragment d’alignement de séquences d’ORF5 de souches de SDRPv de 5 élevages différents. A partir des élevages 1, 4 et 5, on a obtenu de multiples séquences. Les points représentent des positions identiques à la souche de référence : un SDRPv de type 1, Lelystad.
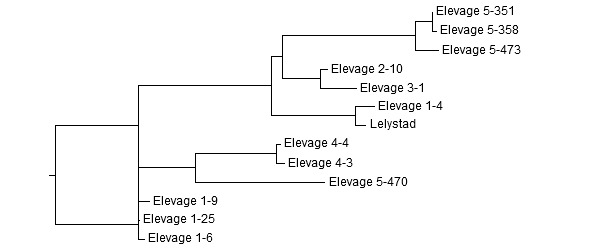
Figure 2 : dendrogramme de séquences ORF5 obtenues de 5 élevages différents. Exemple d’interprétation : dans l’élevage 1 il y a deux souches sans rapport. Trois séquences sont > 99% identiques entre elles, alors que la quatrième est en lien à environ 83%. En revanche, elle est en relation avec le virus Lelystad. Les souches des élevages 2 et 3 sont très liées (98,2% identiques). Deux souches de l’élevage 4 sont très liées (99,5% identiques). Dans l’élevage 5 il y a deux souches distinctes : 3 séquences sont identiques entre elles à > 98% et à 81% avec la quatrième
Tableau 1 : pourcentage d’identité par paire parmi tous les séquences alignées d’ORF5 d’une série d’échantillons de SDRPv de type 1
| 1-4 | 1-6 | 1-9 | 1-25 | 2-10 | 3-1 | 4-4 | 4-3 | 5-351 | 5-358 | 5-470 | 5-473 | Lelystad | |
| *** | 83,5 | 83,3 | 83,7 | 93,2 | 92,6 | 86,1 | 86,0 | 88,1 | 88,0 | 83,2 | 88,3 | 98,7 | 1-4 |
| *** | 99,2 | 99,7 | 84,2 | 83,0 | 86,0 | 86,0 | 81,4 | 81,2 | 86,3 | 81,4 | 82,1 | 1-6 | |
| *** | 99,5 | 84,5 | 83,3 | 85,6 | 85,6 | 81,4 | 81,2 | 86,5 | 81,4 | 81,9 | 1-9 | ||
| *** | 84,5 | 83,3 | 86,0 | 86,0 | 81,7 | 81,5 | 86,8 | 81,7 | 82,2 | 1-25 | |||
| *** | 98,2 | 86,6 | 86,5 | 91,1 | 90,9 | 84,2 | 90,4 | 93,3 | 2-10 | ||||
| *** | 84,4 | 84,2 | 90,2 | 90,0 | 83,3 | 89,7 | 92,9 | 3-1 | |||||
| *** | 99,5 | 82,5 | 82,7 | 90,6 | 82,5 | 84,8 | 4-4 | ||||||
| *** | 82,3 | 82,5 | 90,9 | 82,2 | 84,6 | 4-3 | |||||||
| *** | 99,8 | 81,0 | 98,3 | 88,4 | 5-351 | ||||||||
| *** | 81,2 | 98,2 | 88,2 | 5-358 | |||||||||
| *** | 80,9 | 83,2 | 5-470 | ||||||||||
| *** | 88,8 | 5-473 | |||||||||||
| *** | Lelystad |

Interprétation des séquences de SDRPv
La séquence d’ORF5 a quelques 600 nucléotides. Plusieurs estimations montrent que le taux global de mutation de ce gène est environ de 0,5-1% par an. Les variations sur le taux de changement génétique sont déterminées par différents facteurs non viraux. Le niveau d’immunité spécifique contre le SDRPv des porcs a un impact important sur la réplication et la transmission virale, en exerçant une forte pression inhibitrice qui diminue le nombre de copies. Une charge virale réduite entraîne un taux de transmission plus faible, limitant encore plus la réplication virale et diminuant le taux de changement. Le même virus peut montrer différents taux de changement génétique sous différentes conditions, par conséquent, dans certains cas, on peut observer des taux de changements plus importants ou moins importants que l’intervalle suggéré de 0,5-1% par an.
La question principale dans l’analyse génétique est de savoir si des séquençages sont très en relation (appartiennent à deux variants de la même souche) ou sont indépendantes (appartiennent à deux souches non liées). On accepte communément que deux isolats de SDRPv sont en relation ou non si leur similitude est au-dessus ou en dessous de 97-98%. Il est évident que se baser uniquement sur une différence génétique de 2 ou 3% entre les isolats, sans information supplémentaire, peut donner lieu à des conclusions erronées. Les différences entre les variants d’une même souche circulant dans une population pendant plusieurs années peuvent dépasser ce chiffre. L’interprétation de la relation sera meilleure si on a accès à une information supplémentaire comprenant les dates et les lieux des isolements. Il est très important de comparer les nouvelles séquences de SDRPv avec un large panel de référence qui soit représentatif de l’élevage, du système et de la région, et qui représente aussi la diversité génétique globale.
Le séquençage de l’ADN des chaînes du SDRPv peut indiquer une relation de proximité ou d’indépendance des chaînes (figure 2) mais ne peut pas prévoir ou expliquer la protection immunologique ou les épisodes de SDRP dans les élevages immuns. Il ne permet pas non plus la prévision de l’évolution clinique de l’infection d’une souche en particulier puisque les marqueurs génétiques de virulence n’ont pas encore été identifiés.
Actuellement, il y a beaucoup de projets régionaux en œuvre pour le contrôle ou l’élimination du SDRP, particulièrement aux USA mais aussi en Europe. Avoir une vision complète de la variabilité virale au début du projet de contrôle et d’élimination est essentiel pour un suivi efficace du progrès et de l’efficacité des procédés mis en place et pour identifier les nouveaux virus introduits dans les élevages de la région.