Escherichia coli est présente dans la flore intestinale du porc et, sous certaines circonstances, elle produit des tableaux cliniques de colibacillose, une maladie qui peut se manifester par des processus différents, comme la diarrhée néonatale, la diarrhée post-sevrage, la maladie de l’œdème, une septicémie, entre autres.
Pour un bon diagnostic, une anamnèse exhaustive ainsi qu'une sélection appropriée des échantillons et un complet diagnostic différentiel soutenu par plusieurs techniques sont nécessaires. La simple détection de souches pathogènes ne justifie pas toujours la maladie, car il est habituel de trouver des porteurs asymptomatiques. L'identification de souches significatives en raison de leur virulence et leur différenciation du reste de la flore intestinale habituelle constitue un défi diagnostique pleinement d'actualité, et la PCR en temps réel (qPCR) appliquée à la détection des facteurs de virulence (FV) d'E. coli s'avère un outil puissant pouvant générer des informations précieuses.

À partir de l'extraction des acides nucléiques de différentes matrices biologiques (culture, fèces, intestin ou écouvillonage rectal), les tests qPCR reconnaissent des régions spécifiques du génome d'E. coli codant différents FV. Dans cette étude, chaque test a été conçu pour la détection d'un FV différent (Tableau 1). Une qPCR additionnelle (ECCO) a été utilisée pour confirmer la présence d'E. coli dans les échantillons et pour garantir que toutes les étapes de la technique ont été correctement suivies. La qPCR est à caractère qualitatif mais aussi quantitatif (Image 1), ce qui constitue un avantage considérable par rapport à son prédécesseur, la PCR conventionnelle.
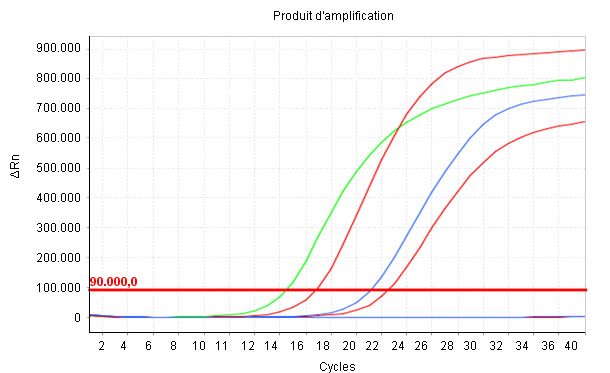
Image 1. Le produit d'amplification est montré en temps réel au fur et à mesure que les cycles thermiques (Cycles) se succèdent, de telle sorte que le niveau de fluorescence enregistré (ΔRn) augmente de manière exponentielle. Le Cq est le nombre de cycles dans lequel la fluorescence commence à être détectée. Plus la valeur Cq est petite, plus la concentration initiale du paramètre étudié dans l'échantillon est élevée.
Dans les processus de colibacillose, les souches virulentes ont été traditionnellement évaluées sur la base d'une culture microbiologique initiale, l'isolement des colonies d'E. coli et la caractérisation ultérieure de leurs FV. L'inconvénient de cette méthodologie est que l'analyse est limitée au nombre d'isolements sélectionnés, et l'exactitude du diagnostic dépend donc de leur représentativité au sein du total de la population d'E. coli dans l'échantillon étudié. Ainsi, les populations non majoritaires d'E. coli qui soient cependant cliniquement significatives risquent de passer inaperçues.
La qPCR permet d'analyser les FV directement sur l'échantillon clinique. Cette méthodologie évite les étapes intermédiaires de culture ainsi que les déterminations respectives sur chacun des différents isolats sélectionnés, ce qui entraîne des économies considérables en temps et en coûts. Compte tenu du caractère quantitatif de cette technique, la relation entre le nombre de copies détecté pour un gène de virulence spécifique et la population totale d'E. coli présente dans l'échantillon peut être déterminée. Ainsi, la possibilité de pouvoir isoler une souche avec une combinaison spécifique de FV pourrait être interprétée.
Cette méthodologie présente certaines contraintes à cause du nombre indéterminé de copies des différents gènes codant chaque FV au sein de populations bactériennes différentes. Cependant, elle permet d'atteindre l'objectif envisagé : évaluer la possibilité de trouver dans l'échantillon une population d'E. coli avec une combinaison particulière de FV.
Voici ci-après un exemple pratique. Face à un cas de suspicion clinique de maladie des oedèmes chez des animaux d'engraissement, quatre intestins grêles appartenant à des animaux malades ont été remis au laboratoire pour déterminer la présence d'E. coli vérotoxigénique. Le pool formé par les quatre intestins a été directement analysé par qPCR, et les valeurs Cq résultantes pour F18, Stx2e et ECCO ont été pratiquement égales (Tableau 1). Ces résultats peuvent être interprétés comme indiquant la présence d'une population largement majoritaire d'E. coli possédant les gènes codant pour F18 et Stx2e, c'est-à-dire, une souche vérotoxigénique. En outre, une valeur faiblement positive pour F4 (Cq = 36,9) a été obtenue, ce qui indique une présence très minoritaire de populations avec ce FV.
Simultanément, une culture microbiologique des échantillons a été réalisée, dans laquelle 12 colonies d'E. coli ont été sélectionnées. Leur comparaison phénotypique a révélé la présence d'une seule souche (Image 2). La caractérisation de celle-ci au moyen de la détermination de ses FV par qPCR a confirmé qu'il s'agissait d'une souche vérotoxigénique potentielle car les gènes codant pour F18 et Stx2e ont été simultanément détectés.
Tableau 1. Résultats qPCR.
| FV | Pool 4 intestins grêles | Souche E. coli isolée |
| F4 | POS (Cq = 36,9) | nég |
| F5 | nég | nég |
| F6 | nég | nég |
| F18 | POS (Cq = 22,28) | POS (Cq = 16,58) |
| F41 | nég | nég |
| LT | nég | nég |
| STa | nég | nég |
| STb | nég | nég |
| Stx2e | POS (Cq = 22,39) | POS (Cq = 17,29) |
| eae | nég | nég |
| ECCO | POS (Cq = 21,1) | POS (Cq = 16,97) |
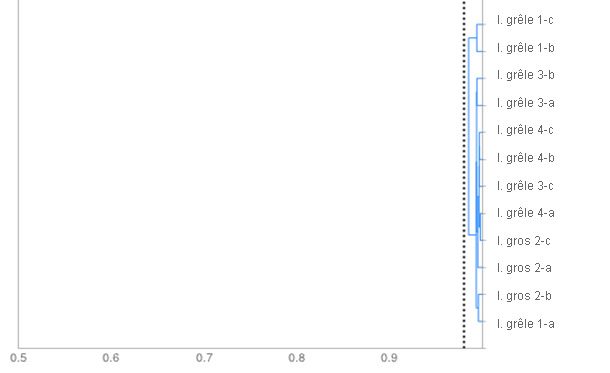

Image 2. Dendogramme. Le degré de similarité entre les isolats d'E. coli est montré en fonction du modèle de fermentation des sucres. Tous les isolats présentent une similarité supérieure au point de coupure établi à 0,98 (pointillé), ce qui permet de conclure qu'il s'agit d'une seule souche.
En conclusion, la qPCR appliquée directement sur les échantillons cliniques peut représenter des économies considérables en ressources et en temps, tout en offrant des informations qui peuvent nous conduire à une interprétation qui apporterait des solutions au diagnostic délicat des colibacilloses porcines.



